标新生物在Nature子刊联合发表文章,开发基于深度学习的PROTACs降解功效预测方法
发布日期:2022-11-24 文章来源:标新生物
近日,标新生物和上海科技大学免疫化学研究所以及信息科学与技术学院联合在国际学术期刊《Nature Communications》上发表题为“DeepPROTACs is a deep learning-based targeted degradation predictor for PROTACs”的研究论文。该文章设计了一个深度神经网络模型,可以根据靶蛋白、E3连接酶和PROTACs的结构预测所设计的PROTACs分子的降解药效。
PROTACs(PROteolysis TArgeting Chimeras)是一种特异性双功能分子,由靶蛋白配体、连接子和E3泛素连接酶配体组成。它通过促进靶蛋白-PROTAC-E3酶三元复合物的形成,驱动泛素从E2泛素结合酶转移至靶蛋白并与表面的赖氨酸共价结合。随后,经泛素标记的靶蛋白被26S蛋白酶体识别并被降解为短肽甚至氨基酸(图1)。
与小分子抑制剂相比,PROTACs显示出多种优越的特性,如能够靶向不可成药的蛋白、可以较为有效的缓解药物的获得性耐药性、对靶标蛋白的亲和力要求低等。然而,鉴于靶蛋白-PROTAC-E3酶三元复合物的实验结构稀少,计算结构难以获得,PROTACs的构效关系并不明确。目前仍无PROTAC理想的理性设计与药效评价计算方法,PROTACs的发现主要凭借药物化学家通过经验设计长短不一、结构各异的连接子将已知的靶蛋白配体和E3酶配体连接,然后使用有机合成、蛋白质免疫印迹分析等实验手段筛选出对目标蛋白有降解效力的PROTACs分子。
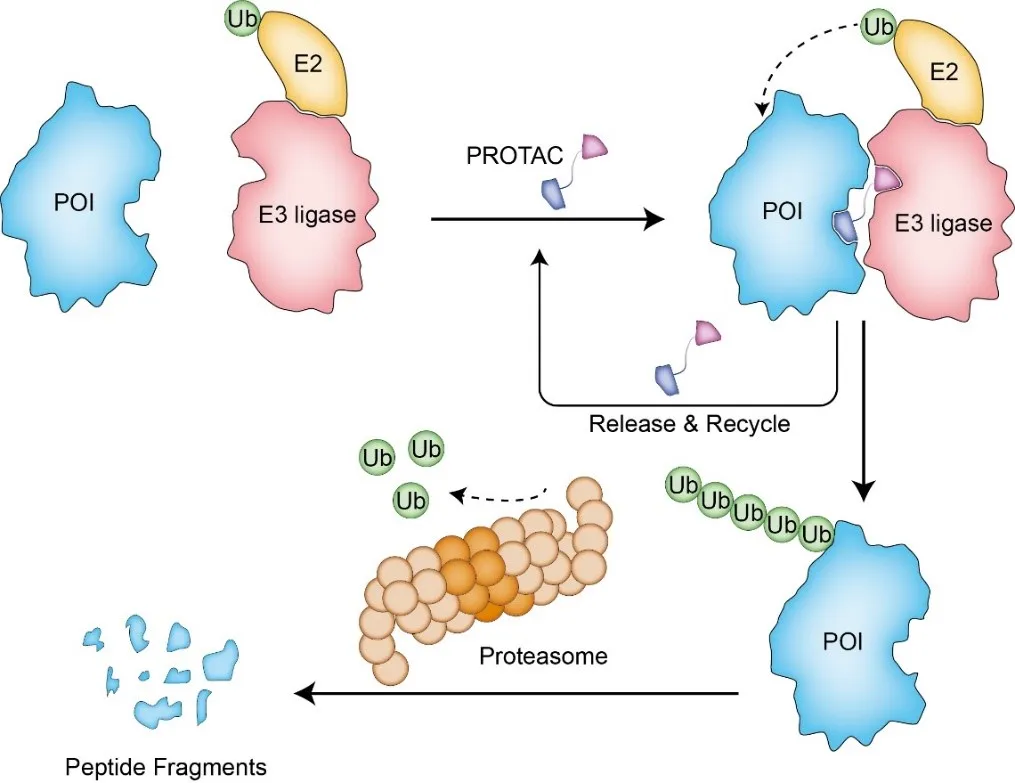
图1:PROTACs对靶蛋白的降解机理
为了有效指导PROTACs的理性设计,特别是连接子的设计,本研究提出了一个以神经网络为基础的深度学习模型DeepPROTACs,用于预测设计出的PROTACs对于靶蛋白的降解功效。用于深度学习的数据主要来源于PROTAC-DB数据库及额外收集的PROTACs数据。DeepPROTACs模型以半降解浓度(DC50)和最大降解水平(Dmax)为依据,将降解率的预测简化为二分类问题。研究团队规避PROTACs三元复合物的复杂模建过程,从已经确定的蛋白-配体结构中提取出五个重要部分,即靶蛋白口袋、E3酶口袋、靶蛋白配体、E3酶配体以及连接子,并使用五个模块分别提取相应特征。对于连接子,DeepPROTACs使用双向LSTM作为特征提取器,其余的模块均使用神经网络进行特征提取。最后使用多层感知机对合并的特征向量进行输出,预测降解功效(图2)。该模型在测试集上的平均预测准确率达到78%左右,ROC曲线下面积(AUROC)达到0.85左右,说明模型的预测性能良好。另外,DeepPROTACs对外部实验集(ER蛋白的PROTACs)和不包含在训练集里的数据(EZH2、STAT3、eIF4E、FLT-3等蛋白的PROTACs)的预测准确率在65% – 80%之间,表明模型具备良好的泛化能力。本研究发布了网络服务器(https://bailab.siais.shanghaitech.edu.cn/services/deepprotacs/)和源代码(https://github.com/fenglei104/DeepPROTACs)以方便读者使用模型进行预测或者更改模型以满足自定义的需求。综上,DeepPROTACs不仅为PROTACs的设计提供了一种高通量筛选方法,而且为AI与药物发现的融合提供了一种研究范式。
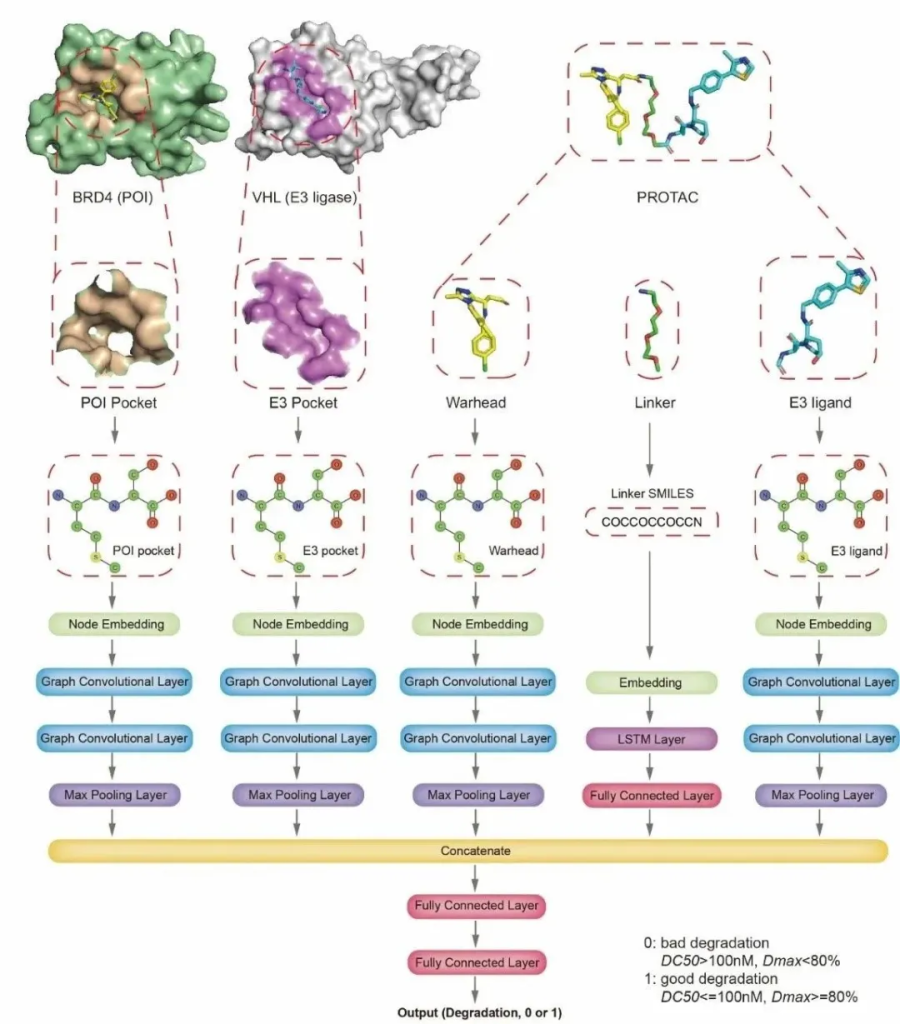
图2:DeepPROTACs模型的网络架构
该文章是标新生物成立以来与学术机构合作在线发表的第五篇通讯单位文章,此篇学术论文的发表标志着标新生物将由传统的药物化学主导的蛋白降解药物设计平台迈入了AI药物设计平台时代,将进一步降低药物研发周期以及药物研发的资金投入,进一步展示了GLUETACS双蛋白降解小分子平台加持AI药物算法助力该领域的快速商业化和产品化的能力。